Metódy štúdie haplotype, diagnostika, choroby
- 1607
- 362
- Blažej Hrmo
A Haplootyp Je to oblasť genómu, ktorá má tendenciu zdediť spolu viacerých generácií; Zvyčajne sa všetko nachádza v rovnakom chromozóme. Haplotypy sú produktom genetickej väzby a zostávajú nedotknuté počas genetickej rekombinácie.
Slovo „haplotipo“ pochádza z kombinácie slova „haploid“ a slovo „genotyp“. „Haploid“ sa vzťahuje na bunky s jednou sadou chromozómov a „genotyp“ hovorí o genetickom zložení organizmu.
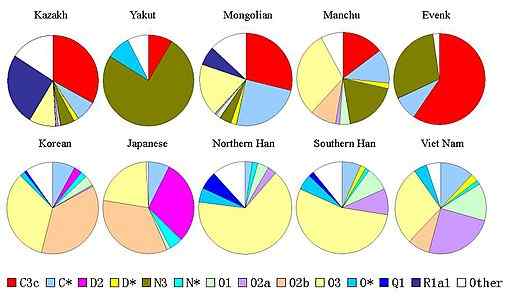
 Schéma distribúcie chromozómových haplotypov a v ázijských populáciách (zdroj: Moogalelod [verejná doména] prostredníctvom Wikimedia Commons) Po definícii môže haplotyp opísať niekoľko génov alebo viac, ktoré sú zdedené spolu na chromozóme z rodičovského alebo môže opísať chromozóm, ktorý je úplne zdedený od rodičov, ako je to v prípade chromozómu a mužov.
Schéma distribúcie chromozómových haplotypov a v ázijských populáciách (zdroj: Moogalelod [verejná doména] prostredníctvom Wikimedia Commons) Po definícii môže haplotyp opísať niekoľko génov alebo viac, ktoré sú zdedené spolu na chromozóme z rodičovského alebo môže opísať chromozóm, ktorý je úplne zdedený od rodičov, ako je to v prípade chromozómu a mužov.
Napríklad, keď haplotypy zdieľajú gény pre dva rôzne fenotypické znaky, ako je farba vlasov a farba očí, jednotlivci, ktorí majú gén farby vlasov, budú mať aj druhý gén pre farbu očí.
Haplotypes sú jedným z najpoužívanejších nástrojov na štúdium genealógie, na sledovanie pôvodu chorôb, aby sa charakterizovala genetická variabilita a fylogeografia populácií rôznych typov živých bytostí.
Existuje niekoľko nástrojov na štúdium halootypov, jedným z najpoužívanejších dnešných je “Mapa haplotypu„(Hapmap), čo je webová stránka, ktorá umožňuje určiť, aké sú segmenty genómu, ktoré sú haplotypmi.
[TOC]
Metódy štúdie
Haplotypy predstavujú príležitosť porozumieť dedičstvu génov a ich polymorfizmu. S objavom techniky „polymerázová reťazová reakcia“ (PCR, angličtina “Polymerická reťazová reakcia“) V štúdii haplotypov široko postupoval.
V súčasnosti existuje veľa metodík na štúdium haplotypov, niektoré z najdôležitejších sú:
Sekvenovanie DNA a detekcia polymorfizmu s jedným nukleotidom (SNP)
Vývoj technológií sekvencovania novej generácie predstavoval veľký skok na štúdium haplotypov. Nové technológie umožňujú variácie až do jednej nukleotidovej bázy v konkrétnych oblastiach haplotypu.
Môže vám slúžiť: rekombinantná DNA: technika, aplikácie a základyV bioinformatike sa pojem haplotyp tiež používa na označenie dedičstva jediných nukleotidových polymorfizmov (SNP) v sekvenciách DNA.
Kombinácia bioinformatických programov s detekciou haplotypov pomocou sekvenovania novej generácie môže byť presne identifikovaná pozícia, náhrada a účinok zmeny každého základu na genóme populácie.
Mikrosatelity (SSR)
Mikrosatelity alebo SSR odvodzujú svoje meno z angličtiny „Simplementovať opakovanie a Krátke tandemové opakovanie„. Jedná sa o krátke nukleotidové sekvencie, ktoré sa postupne opakujú v oblasti genómu.
Je bežné nájsť mikrosatelity vo vnútri haplotypov nekódujúcich.
Na detekciu nekonečných haplotypov boli vyvinuté mikrosatelitné molekulárne markery, od sexuálnych rastlín, ako je papája (Carica Papaya) Až do detekcie ľudských chorôb, ako je anémia falciform.
Polymorfizmy zosilnených fragmentov (AFLP)
Táto technika kombinuje amplifikáciu s reakciami PCR s trávením DNA s dvoma rôznymi reštrikčnými enzýmami. Táto technika detekuje polymorfné lokusy v haplootypoch podľa rôznych rezných miest v sekvencii DNA.
Aby sme lepšie ilustrovali túto techniku, predstavme si tri tkaninové fragmenty rovnakej dĺžky, ale nakrájame na rôzne miesta (tieto fragmenty predstavujú tri fragmenty zosilnených haplotypov pomocou techniky PCR).
V čase, keď je tkanina rezaná, získa sa veľa kusov rôznej veľkosti, pretože každá látka sa rozreže na rôzne miesta. Pri objednávaní fragmentov podľa typu látky, z ktorej pochádzajú, môžeme pozorovať, kde sa nachádzajú rozdiely medzi látkami alebo haplotypmi.
Diagnostika a choroby
Dôležitou výhodou genetickej štúdie haplotypov je to, že zostávajú takmer neporušené alebo nezmenené pre tisíce generácií, čo umožňuje identifikáciu vzdialených predkov a každú z mutácií, s ktorými jednotlivci prispievajú k rozvoju chorôb.
Môže vám slúžiť: fenotypické variácieHaplotypy v ľudskosti sa líšia v závislosti od rás a od tejto kopce sa gény detegovali v haplotypoch, ktoré spôsobujú vážne choroby v každom z ľudských rás.
V projekte Hapmap Zahrnuté sú štyri rasové skupiny: Európska, nigérijská Yoruba, čínsky Han a Japonec.
Týmto spôsobom projekt Hapmap Môže pokrývať rôzne skupiny populácií a sledovať pôvod a vývoj mnohých dedičných chorôb, ktoré ovplyvňujú každú zo štyroch pretekov.
Jednou z chorôb, ktoré sa najčastejšie diagnostikuje pomocou haplotypovej analýzy, je falciformná anémia u ľudí. Toto ochorenie je diagnostikované sledovaním frekvencie afrických haplotypov do populácie.
Identifikácia afrických haplotypov v populáciách, ktorá bola pôvodom z Afriky, vám umožní ľahko sledovať ľudí, ktorí majú mutáciu v genetickej sekvencii pre beta globíny erytrocytov vo forme Hoz (charakteristika patológie).
Príklady
S haplotypmi fylogenetickými stromami, ktoré predstavujú evolučné vzťahy medzi každým z haplotypov, ktoré sa nachádzajú vo vzorke homológnych molekúl DNA alebo toho istého druhu, sa konštruujú v oblasti, ktorá má malú alebo žiadnu rekombináciu.
Jednou z najviac študovaných vetiev prostredníctvom haplotypov je vývoj imunitného systému ľudí. Haplotypes, ktoré kódujú receptor typu Toll (kľúčová súčasť vrodeného imunitného systému), boli identifikované pre neandertálsky a genóm Denisovanos.
To im umožňuje sledovať, ako sa genetické sekvencie zmenili v populáciách „moderných“ ľudí vzhľadom na haplotypové sekvencie, ktoré zodpovedajú „predkom“ ľudí.
Budovanie siete genetických vzťahov z mitochondriálneho haplotypu.
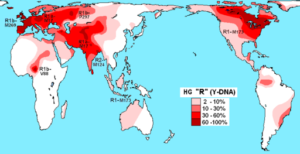 Distribúcia haploskupín R (DNA-Y) v natívnych populáciách (zdroj: maucioni [CC po 3.0 (https: // creativeCommons.Org/licencie/o/3.0)], cez Wikimedia Commons) Diverzita haplotypov sa používa na sledovanie a štúdium genetickej diverzity zvierat vychovaných v zajatí. Tieto techniky sa používajú špeciálne pre náročné druhy na monitorovanie voľne žijúcich živočíchov.
Distribúcia haploskupín R (DNA-Y) v natívnych populáciách (zdroj: maucioni [CC po 3.0 (https: // creativeCommons.Org/licencie/o/3.0)], cez Wikimedia Commons) Diverzita haplotypov sa používa na sledovanie a štúdium genetickej diverzity zvierat vychovaných v zajatí. Tieto techniky sa používajú špeciálne pre náročné druhy na monitorovanie voľne žijúcich živočíchov.
Zvieracie druhy, ako sú žraloky, vtáky a veľké cicavce, ako sú jaguáre, slony, sa neustále geneticky hodnotia prostredníctvom mitochondriálnych haplotypov na monitorovanie genetického stavu populácií v zajatí v zajatí.
Odkazy
- Bahlo, m., Stankovich, J., Rýchlosť, t. P., Rubio, J. P., Burfoot, r. Klimatizovať., & Noha, s. J. (2006). Detapovanie genómu široké zdieľanie haplotypov pomocou údajov SNP alebo mikrosatelitových haplotypových údajov. Human Genetics, 119 (1-2), 38-50.
- Dannemann, m., Andrés, a. M., & Kelso, J. (2016). Introgress neandertálskych haplotypov podobných haplotypom prispieva k adaptívnym variáciám v ľudských mýtnych príjemcoch. The American Journal of Human Genetics, 98 (1), 22-33.
- Od Vries, h. G., Van der Meulen, m. Do., Rozen, r., Halley, D. J., Scheffer, h., Leo, P.,... a ty meerman, g. J. (Devätnásť deväťdesiat šiestich). Identita haplotypu medzi jednotlivcami, ktorí zdieľajú alelu mutácie CFTR. Human Genetics, 98 (3), 304-309
- Prehltnúť, m. Do., Opustenie, a. L., Christiansen, f. Tón., Witt, C. Siež., Abrahám, L. J., & Dawkins, R. L. (1992). Haplotypy predkov: populácia Conserva MHC haplotypy. Human Immunology, 34 (4), 242-252.
- Fellows, M. R., Hartman, T., Hermelin, D., Landau, G. M., Rosamond, f., & Rozenberg, L. (2009, jún). Inferencia haplotypu obmedzená pomocou hodnoverných údajov haplotypu. V ročnom sympóziu o zhode kombinatorického vzoru (str. 339-352). Springer, Berlín, Heidelberg.
- Gabriel, s. B., Schaffner, s. F., Nguyen, h., Moore, J. M., Roy, J., Blumenstiel, b.,... & Liu-Cordero, s. N. (2002). Štruktúra haplotypových blokov v ľudskom genóme. Science, 296 (5576), 2225-2229.
- Medzinárodné konzorcium HapMap. (2005). Mapa haplotypu ľudského genómu. Nature, 437 (7063), 1299.
- Wynne, r., & Wilding, C. (2018). Mithochondriálna diverzita haplotypu DNA a pôvod žralokov v zajatí pieskových tigrov (Carcharias Taurus). Journal of Zoo and Aquarium Research, 6 (3), 74-78.
- Yoo a. J., Tang, J., Kaslow, r. Do., & Zhang, K. (2007). Inferencia haplotypu pre súčasné údaje o genotype s použitím predvolených haplotypov a haplotypových vzorov. Bioinformatics, 23 (18), 2399-2406.
- Mladý, n. Siež. (2018). Aplastická anémia. The New England Journal of Medicine, 379 (17), 1643-1656.
- « Hollandské dedičské charakteristiky, funkcie génov, degenerácia
- Lentén väčšie vlastnosti, biotop, vlastnosti, starostlivosť »

